Planter genkender sygdomsfremkaldende og nyttige mikroorganismer
Forskere fra Aarhus Universitet har i samarbejde med nationale og internationale eksperter afsløret nye grundlæggende træk i de biomolekylære interaktioner, der sætter planter i stand til at identificere og reagere hensigtsmæssigt på mikroorganismer. De nye resultater giver en bedre forståelse af de mekanismer, der styrer planters evne til at interagere med nyttige mikroorganismer og samtidig være modstandsdygtige over for sygdomsfremkaldende bakterier og svampe. Dette vil kunne få betydning for fremtidens bæredygtige landbrug, hvor sprøjtemidler i stigende grad søges erstattet med nyttige mikroorganismer.

Planters rødder er omgivet af tusindvis af bakterier og svampe, der lever i jorden og på røddernes overflade. For at overleve i dette mangfoldige miljø anvender planterne avancerede detektionssystemer for at skelne sygdomsfremkaldende mikroorganismer fra nyttige såvel som harmløse mikroorganismer.
Her spiller såkaldte kitin-molekyler og modificerede kitin-molekyler, der stammer fra mikroorganismerne, en vigtig rolle, da de registreres af plantens overvågningssystem. For eksempel opbygger bælgplanter et forsvar mod sygdomsfremkaldende mikroorganismer som en reaktion på simple kitin-molekyler.
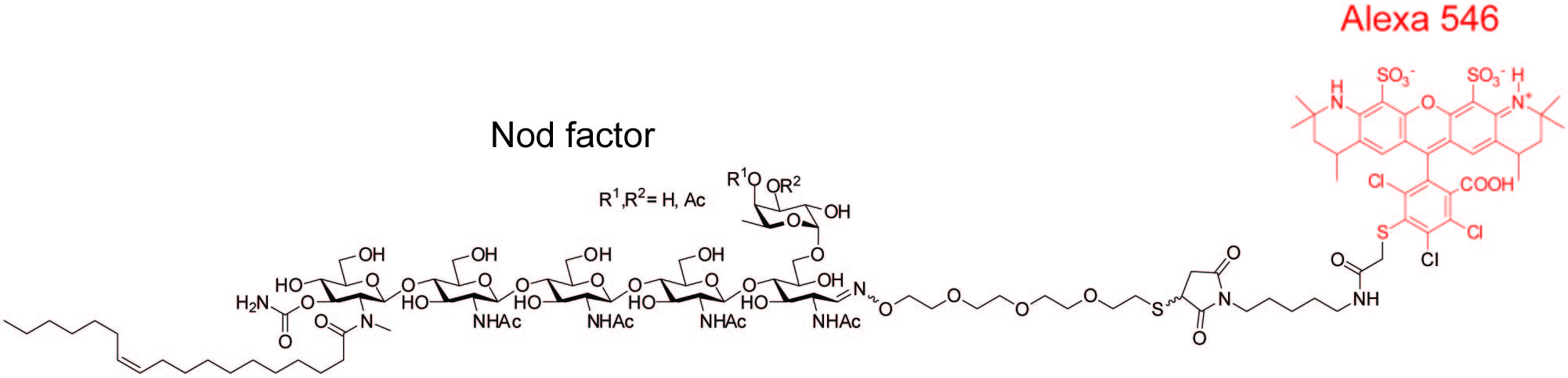
Når planten derimod detekterer et modificeret kitin-molekyle udskilt fra rhizobiumjordbakterier, såkaldte Nod-faktorer, fører dette til dannelse af nye organer i form af ”rodknolde”, som tillader rhizobium-bakterierne at invadere og etablere sig i disse symbiotiske organer. Rhizobium-bakterierne producerer derefter kvælstof til planten.
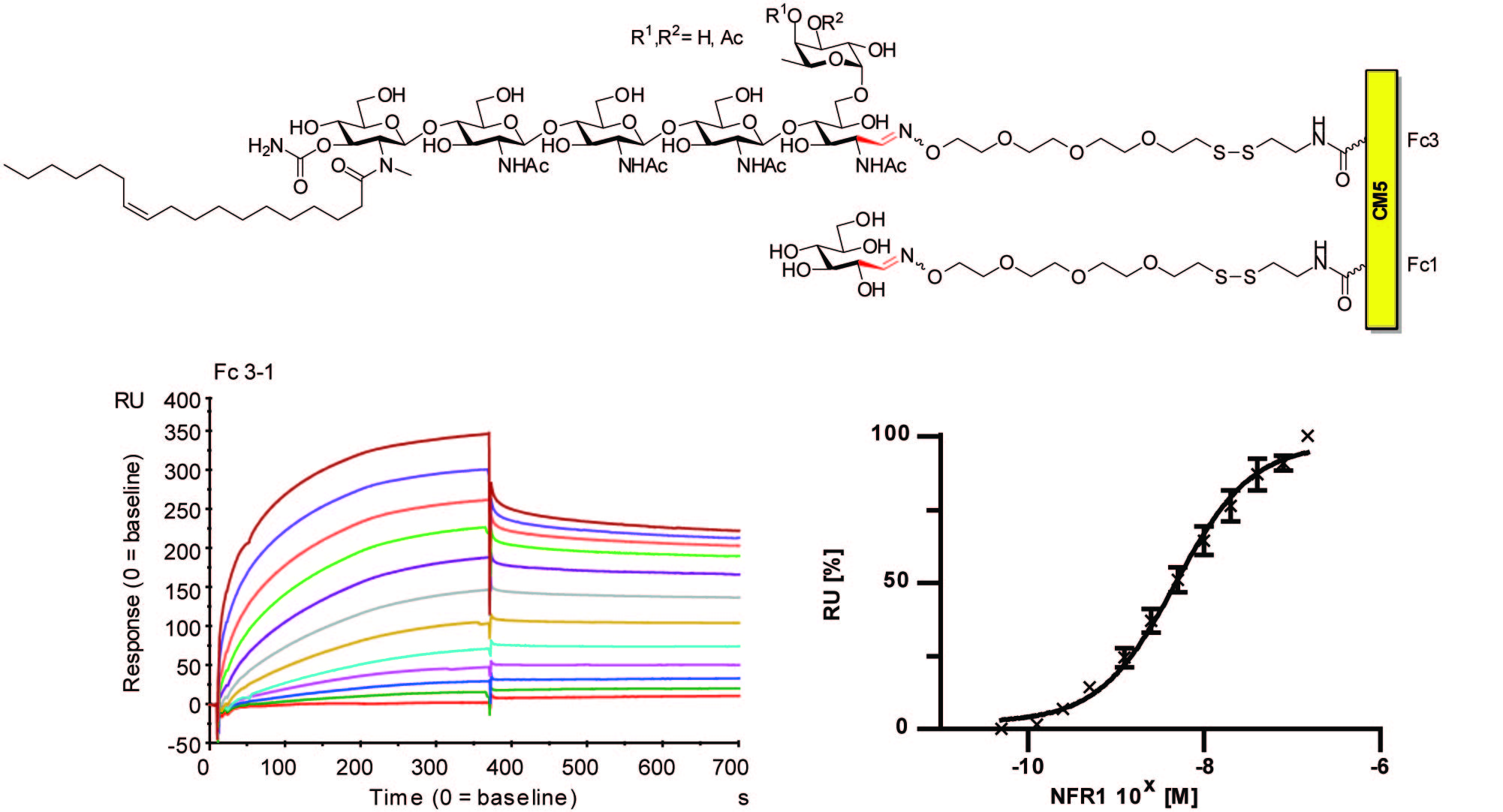
Planters detektion af ligander, såsom kitin og modificerede Nod-faktorer, sker gennem proteinreceptorer, der er lokaliseret i cellernes overflade. Forskningen ved Danmark Grundforskningsfonds Center for Kulhydratgenkendelse og Signallering (CARB) har nu vist, at ligand-genkendelse ved direkte binding af Nod-faktor molekyler er et vigtigt skridt i den process, der fører til udvikling af rodknolde i bælgplanter.
Receptor-bindingen sker ved nano-molære koncentrationer af Nod-faktor, hvilket er sammenligneligt med de biologisk relevante koncentrationer, hvor Nod-faktor har in vivo aktivitet. Simple kitin-ligander binder derimod kun til recepetorerne med lav affinitet ved mikromolære koncentrationer. Receptor-genkendelsen er således bestemt af ligandens opbygning og struktur, og ligand-bindings-affiniteten på forskellige receptorer kan derfor bestemme, hvilken responsmekanisme der aktiveres i planten.
Ved at inddrage flere forskellige tværfaglige discipliner, såsom avanceret biokemi, kemoselektiv kemi og mikrobiel genetik, var det muligt for forskerne at undersøge de molekylære mekanismer, der er involveret i at skelne mellem Nod-faktor-molekyler, der udskilles fra rhizobium, og kitin der udskilles fra sygdomsfremkaldende mikroorganismer.
En af udfordringerne var at oprense receptorproteiner, som er til stede i meget små mængder i rødder af bælgplanten japansk kællingetand (Lotus japonicus), der blev brugt som modelplante. Denne udfordring blev løst ved at udtrykke receptorerne i forskellige plante-baserede systemer og oprense dem fra membranfraktioner.
En anden udfordring var etableringen af bindingsassays med kulhydrat-ligander. Dette blev løst ved kemisk modifikation af Nod-faktor og kitin ved hjælp af kemoselektiv kulhydrat kemi.
Forskerne bag de resultater, som netop er blevet offentliggjort i det internationale tidsskrift PNAS, er tilknyttet Danmark Grundforskningsfonds Center for Kulhydratgenkendelse og -Signalering på Institut for Molekylær Biologi og Genetik ved Aarhus Universitet, Kemisk Institut ved Københavns Universitet og Institut for Mikrobiologi og Immunologi ved Otago Universitet (New Zealand).
Link til den videnskabelige artikel i PNAS: Legume receptors perceive the rhizobial lipochitin oligosaccharide signal molecules by direct binding
Angelique Broghammera,b, Lene Krusella,b, Mickaël Blaisea,b, Jørgen Sauera,c, John T. Sullivana,d, Nicolai Maolanona,c,
Maria Vinthera,b, Andrea Lorentzene, Esben B. Madsena,b, Knud J. Jensena,c, Peter Roepstorffe, Søren Thirupa,b, Clive W. Ronsona,d, Mikkel B. Thygesena,c, and Jens Stougaarda,b
aCentre for Carbohydrate Recognition and Signalling;
bDepartment of Molecular Biology and Genetics, Aarhus University, DK-8000 Aarhus, Denmark;
cDepartment of Chemistry, Faculty of Science, University of Copenhagen, DK-1871 Frederiksberg C, Denmark;
dDepartment of Microbiology and Immunology, University of Otago, Dunedin 9054, New Zealand;
eDepartment of Biochemistry and Molecular Biology, University of Southern Denmark, DK-5230 Odense M, Denmark
Kontakt
Professor Jens Stougaard
Leder af Danmark Grundforskningsfonds Center for Kulhydratgenkendelse og -Signalering (CARB)
Institut for Molekylærbiologi og Genetik, Aarhus Universitet
stougaard@mb.au.dk - 6020 2649
Tekst: Jens Stougaard og Lisbeth Heilesen
Oversættelse fra engelsk til dansk: Lisbeth Heilesen
1. november 2012
