Nyopdaget RNA nedbrydningsvej i menneskets cellekerner
Genomer transskriberes til et hav af forskelligt RNA. Dette materiale er ikke alt sammen anvendeligt, hvorfor målrettet nedbrydning er essentielt for at opretholde cellulært liv. En nyligt opdaget RNA nedbrydningsvej i menneskets cellekerner er med til netop det.
Vores genomer er hyperaktive, idet de producerer en række forskellige transskripter. Hovedbestanddelen af denne såkaldte ’pervasive’ transskription består af ikke-kodende RNA’er, hvoraf mange nedbrydes hurtigt efter deres produktion. Så hvorfor dannes sådanne ’spøgelsesagtige’ RNA’er i det hele taget? I betragtning af deres normalt korte levetider, er det usandsynligt, at alle er funktionelle. Pervasiv genomaktivitet kan imidlertid være vigtig for andre regulatoriske aspekter af genekspression eller eventuelt spille en rolle som et evolutionært reservoir til skabelse af nye funktionelle transskriptionsenheder. Uanset hvad, så skal mange RNA’er nedbrydes hurtigt, så de ikke forårsager en katastrofal ophobning og derved forstyrrer og forsinker andre vitale cellulære processer.
RNA’er, der nedbrydes, er forskellige – korte eller lange, umodne eller modne – så hvordan bliver de identificeret? Ustabile RNA’er nedbrydes ofte i eukaryote kerner af ribonukleolytiske RNA exosome komplekser, som er velbevarede fra gær til menneske. Til korrekt targetering af dets substrater, udnytter RNA exosomet så-kaldte adapterkomplekser, der garanterer en målrettet fjernelse af de tilsigtede molekyler.
For nogle år opdagede og karakteriserede forskere i Torben Heick Jensens laboratorium ved Institut for Molekylærbiologi og Genetik, Aarhus Universitet, en nuklear exosome adapter, kaldet ’Nuclear EXosome Targeting (NEXT) complex’. NEXT rekrutterer RNA exosomet til korte og primært uforarbejdede RNA’er.* Disse fund gjorde det klart, at der muligvis fandtes en anden adapter, som kunne være målrettet længere og mere forarbejdede RNA’er. En enkel, men effektiv, idé, som førte til den nye opdagelse af ??den såkaldte ’PolyA exosomet Targeting (PAXT)’ forbindelse.
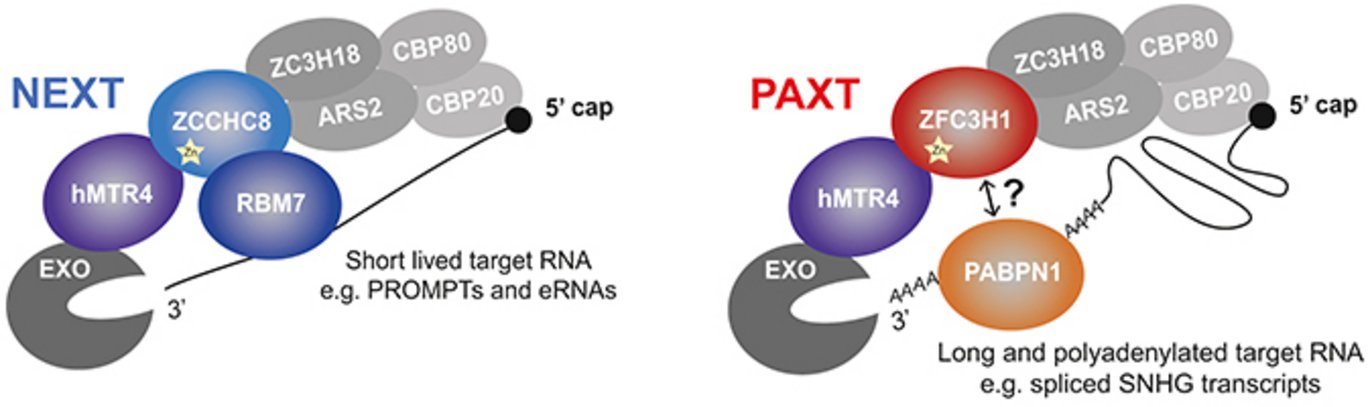
PAXT’s protein sammensætning ligner i nogen grad NEXT’s, idet den også huser RNA-helicasen hMTR4, som er den grundlæggende og direkte fysiske forbindelse til RNA exosomet (se Figur). Desuden huser PAXT et zinkfinger-protein, ZFC3H1 (mens NEXT huser zinkfingerproteinet ZCCHC8) og det polyA-hale-bindende protein PABPN1 (NEXT huser det RNA-bindende protein RBM7), der sikrer PAXT’s genkendelse af hovedsageligt polyadenylerede transskripter.
Trods disse ligheder kan PAXT og NEXT ikke interagere med RNA exosomet samtidigt. At PAXT’s og NEXT’s nedbrydningsveje er funktionelt distinkte blev ligeledes vist ved high-throughput RNA-sekvensanalyse. Her blev især PAXT afsløret i at være målrettet polyadenylerede transskripter, der også er betydeligt længere end de klassiske NEXT substrater. Udover en sådan evne til at ’overvåge’ modningsstatus af nuklear RNA, synes både PAXT og NEXT at være i stand til at detektere cappede RNA’er i kraft af deres fysiske forbindelser til det cap-bindende kompleks (CBC – Cap-Binding Complex) (se Figur). Forskningsgruppen forsøger nu at afsløre andre biokemiske aspekter, der adskiller PAXT og NEXT substrater, ligesom de også forsøger at finde flere uopdagede RNA exosome adaptorer.
De nye resultater, der primært er udført af postdoc Nicola Meola og postdoc Michal Domanski samt ph.d.-studerende Evdoxia Karadoulama – i samarbede med forskere fra Københavns Universitet og Syddansk Universitet – er offentliggjort i Molecular Cell.
*Lubas et al. Mol. Cell 2011; Andersen et al. Na.t Struct. Mol. Biol. 2013; Lubas et al. Cell Reports 2015.
Mere information
Professor Torben Heick Jensen
Institut for Molekylærbiologi og Genetik
Centre for mRNP Biogenese og Metabolisme
Aarhus Universitet
thj@mb.au.dk - mobil: 6020 2705
