3D-visualisering afslører RNA’s detaljerede foldningsproces
I en banebrydende udvikling har forskere fra USA og Danmark med succes fanget 3D-billeder af individuelle RNA-nanopartikler midt i deres foldningsproces. Ved hjælp af en avanceret elektronmikroskopi-teknik har forskerne opnået ny indsigt i den komplekse foldningsdans, der leder til RNA-molekylernes endelige form
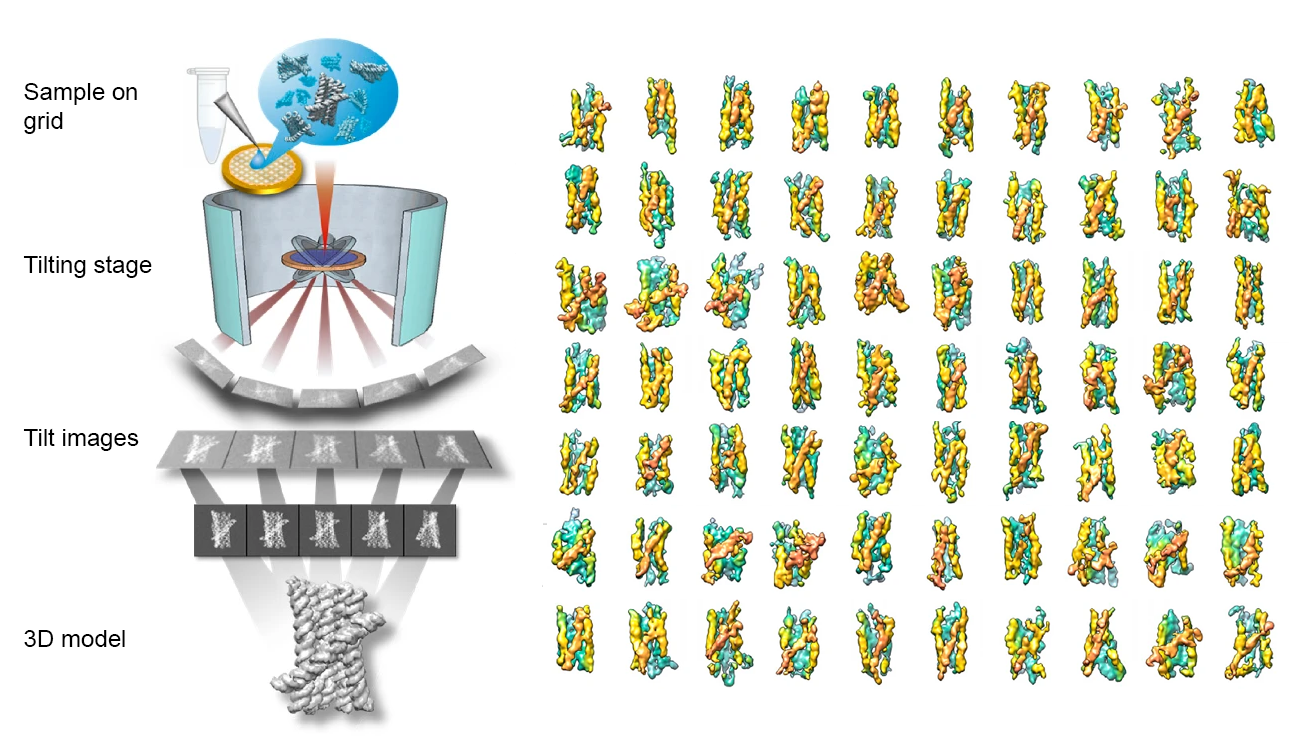
Fleksibiliteten af RNA gør det notorisk udfordrende at studere, da dets struktur kan ændre sig i mange forskellige former afhængigt af de omgivende forhold. Traditionelle metoder til at studere RNA, såsom cryo-elektronmikroskopi (cryo-EM) og single-particle averaging (SPA)-analyse, kræver et gennemsnit af data fra tusindvis af udvalgte molekyler med fælles struktur, hvilket gør det svært at fange de unikke former for individuelle RNA-molekyler.
I et studie publiceret i Nature Communications har forskere fra Molecular Foundry ved Lawrence Berkeley National Laboratory og Interdisciplinary Nanoscience Center og Institut for Molekylærbiologi og Genetik ved Aarhus Universitet undersøgt foldningsprocessen for fleksible RNA-molekyler. De anvendte en innovativ teknik, der kan studere 3D-formen af individuelle molekyler uden at bruge et gennemsnit. Denne teknik bygger på avanceret Individual-Particle cryo-Electron Tomography (IPET), en specialiseret tilgang, der fokuserer på dannelse af 3D-billeder af enkelte molekyler fra prøver bevaret med cryo (se figur og faktaboks nedenfor). Tidligere er denne teknik blevet brugt til at undersøge, hvordan nukleosomer folder DNA og fremkalder faseovergange.
Historisk set mente forskere, at det var umuligt at opnå 3D-billeder fra et enkelt molekyle grundet svage signaler. "Det blev betragtet som en blindgyde-metode siden 1970'erne," siger Gang Ren, en forsker ved Molecular Foundry, der ledte forskningen sammen med Ebbe Andersen fra Aarhus Universitet.
I det nuværende studie brugte forskerne IPET til at undersøge RNA-origami – kunstigt strukturerede RNA-molekyler, der er designet til at folde sig i specifikke stukture på nanoskala. Ebbe Andersen og kolleger havde tidligere anvendt cryo-EM SPA-metoden til at studere 3D-strukturen af RNA-origami, men selve foldningsprocessen forblev uopklaret. Med IPET kunne forskerne fange et øjebliksbillede af RNA's foldningslandskab ved at observere molekyler i forskellige stadier af foldning, fra umodne tilstande til deres optimale form. Forskerne var i stand til at identificere en foldningsfælde og et skift til en mere kompakt form, hvilket gjorde det muligt at skabe en "film," der viser RNA's dynamiske foldningsproces (se video).
Videoen viser processen hvor man fokuserer på et enkelt molekyle for at afsløre dets 3D-form ved hjælp af IPET-metoden. Derefter vises analysen af 120 partikler samt en animation, der illustrerer den dynamiske og komplekse foldningsproces af RNA-origami-partikler.
"IPET-teknikken giver os et mere dynamisk syn på molekylærverdenen. Det er vores håb, at denne indsigt vil gøre det muligt for os at designe mere effektive RNA-vacciner og dynamiske sensorer til molekylær medicin," forklarer Ebbe Andersen.
Studietype:
Eksperimentel Molekylærbiologi
Ekstern finansiering:
Projektet har modtaget støtte fra følgende forskningsbevillinger: US National Institutes of Health: R01HL115153, R01GM104427, R01MH077303 og R01DK042667 (G.R., J.L. og M.Z.), Danmarks Frie Forskningsfond: 9040-00425B (E.S.A. og E.K.S.M.), Canadian Natural Sciences and Engineering Research Council: 532417 (E.K.S.M.), European Research Council (ERC) Consolidator Grant: 683305 (E.S.A., C.G., E.K.S.M.) og Novo Nordisk Fondens Ascending Investigator Grant: 0060694 (E.S.A. og C.G.)
Interessekonflikter:
Forfatterne erklærer ingen interessekonflikter.
Link til den videnskabelige artikel:
"Non-averaged single-molecule tertiary structures reveal RNA self-folding through individual-particle cryo-electron tomography" af Jianfang Liu, Ewan K. S. McRae, Meng Zhang, Cody Geary, Ebbe Sloth Andersen og Gang (Gary) Ren
https://www.nature.com/articles/s41467-024-52914-1
Kontaktoplysninger:
Staff Scientist Gang (Gary) Ren
The Molecular Foundry
Lawrence Berkeley National Laboratory
Email: gren@lbl.gov
Lektor Ebbe Sloth Andersen
Interdisciplinary Nanoscience Center
Institut for Molekylærbiologi og Genetik
Aarhus Universitet
Email: esa@inano.au.dk
