Forskere bestemmer første struktur af protein, som opretholder cellemembranen
Ved hjælp af ny banebrydende elektronmikroskopi har forskere fra Aarhus Universitet som de første i verden bestemt strukturen af en såkaldt lipid-flippase. Opdagelserne giver en bedre forståelse af grundprincipper for, hvordan celler fungerer og holder sig friske, og kan på sigt øge vores kendskab til neurodegenerative sygdomme som Alzheimers.
Alle celler er omgivet af en cellemembran, som afgrænser og beskytter cellens indre fra omverdenen. Membranen består af to lag af fedtstoffer, som kaldes lipider, og fordelingen af specifikke typer lipider mellem de to sider af membranen har stor betydning for, at cellen kan holde sig sund og rask og agere dynamisk. Lipid-flippaser er et membranprotein og sidder derfor selv i cellens membraner, og de har stor betydning for membranens egenskaber, hvor de flytter fedtstoffer fra cellemembranens ydre til det indre lag.
Forskerne har undersøgt lipid flippasen Drs2/Cdc50, som findes i gærceller, hvor den flittigt sørger for at transportere lipider fra den ene til den anden side af cellemembranen. Lignende proteiner findes i mange varianter hos mennesker, hvor de også er knyttet til meget aktive processer i hjerneceller og dermed også af mekanismer, der er centrale for neurologiske sygdomme og demens, f.eks. Alzheimers sygdom.
Et protein består groft sagt af en lang kæde af aminosyrer, som folder sammen til en tredimensionel struktur, der er afgørende for deres funktion. Den eksperimentelle bestemmelse af proteinmolekylers 3D-strukturer kræver avancerede teknikker og er ofte en meget stor udfordring og ikke mindst for membranproteiner, som er yderst vanskelige at håndtere.
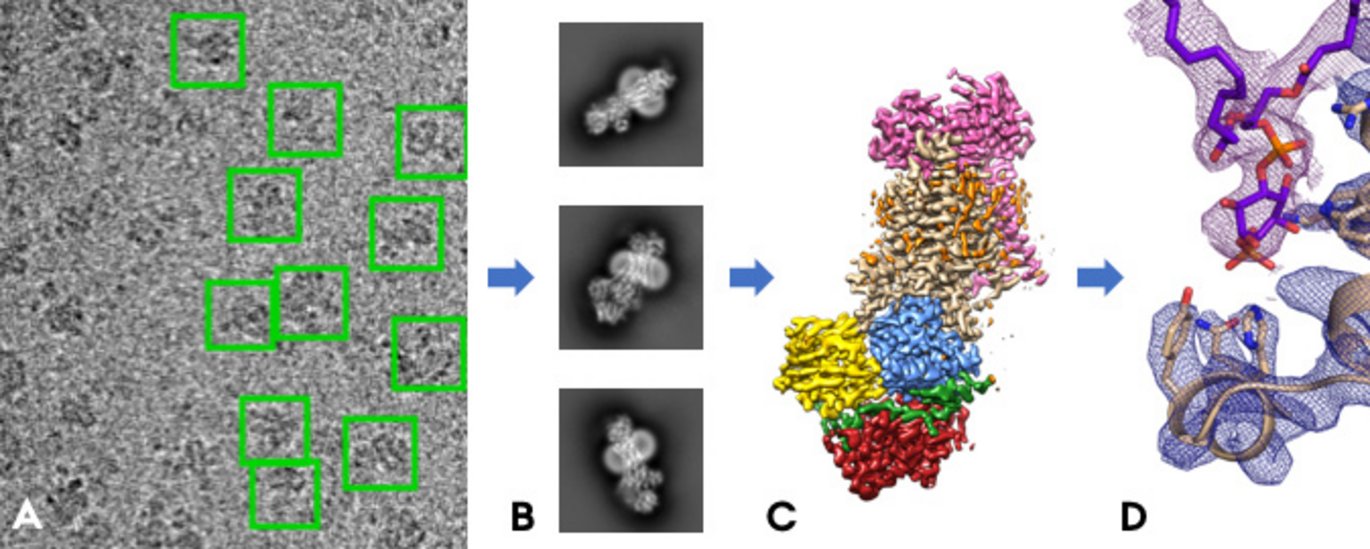
Proteiner er for små til at man kan tage almindelige fotos af dem, men med teknologien cryo-elektronmikroskopi er det lykkedes en gruppe forskere fra professor Poul Nissens forskningsgruppe at bestemme den tredimensionelle struktur af Drs2/Cdc50 ved at tage mange hundredetusinde ”elektronfotografier” af molekylet. Det er første gang, nogen har bestemt en struktur fra denne vigtige familie af proteiner, lipid-flippaserne, og resultaterne er netop blevet publiceret i det verdensførende tidsskrift Nature.
Video of Lipid-mediated aktivering af Drs2p-Cdc50p (Jesper Lykkegaard Karlsen/AU) -(se filmen nederst på siden)
Kulmination af over 10 års arbejde
Med den nylige publikation kulminerer over 10 års målrettet arbejde fra en række studerende og forskere fra Poul Nissens forskningsgruppe, som også har involveret samarbejdspartnere på Université Paris-Saclay og Max Planck Institut for Biofysik i Frankfurt. ”Det er fedt at have været en del af projektets slutspurt, se resultaterne falde på plads og pludselig kunne forklare observationer, som lå flere år tilbage, og som kigger frem i helt nye indsigter,” siger ph.d.-studerende Milena Timcenko, som har arbejdet med bestemmelsen af Drs2/Cdc50’s struktur de sidste fire år sammen adjunkt Joseph Lyons.
Joseph Lyons tilføjer: ”Det er utroligt endelig at se disse membranproteinstrukturer for første gang. Nu begynder det rigtig sjove med at samle det store billede af, hvordan disse transportproteiner fungerer. Det har været en fantastisk udvikling at være med til.”
Cryo-elektronmikroskopi indebærer at proteinerne køles ned til -170 °C i et ultratyndt lag, hvorefter de ”gennemlyses” med en kraftig stråle af elektroner og danner små skygger på en billedchip.
”Det er lidt ligesom, når man tager et røntgenbillede på sygehuset, bare med elektroner i stedet for røntgenstråler og med en forstørrelse på næsten 150.000 gange,” fortæller Milena Timcenko. ”Proteinmolekylerne skal helst ligge helt stille, for at billederne bliver skarpe, samtidig med at de kolde temperaturer beskytter proteinerne fra at blive ødelagt af den kraftige elektronstråle, som udløses af en spænding på 300.000 volt. Det er derfor, vi køler dem ned. Kunsten er at fryse prøverne hurtigt nok til, at der ikke dannes iskrystaller, og i et lag der er tyndt nok til at proteinerne ikke skygger for hinanden. Det er vi efterhånden blevet ret gode til her i Aarhus,” slutter Milena Timcenko.
Resultaterne vækker stor international interesse
Ved at kombinere tusindvis af billeder, hvor proteinet observeres i alle mulige tilfældige retninger, kan man sammensætte et 3D-billede, som gør det muligt at bygge en model af proteinmolekylet, atom for atom, tusinder af atomer. Modellen bidrager til den grundlæggende forståelse af, hvordan lipid-flippaser fungerer, og den kan derfor forklare nogle af de sygdomme, der opstår som følge af mutationer i denne familie af proteiner. De betyder nemlig, at der er sket små ændringer i 3D strukturen, og det kan forskerne nu beskrive effekten af.
Det er første gang en helt ny membranproteinstruktur bestemmes med cryo-elektronmikroskopi i Danmark, og resultaterne vækker stor international interesse. Arbejdet med de store datasæt er blevet muliggjort af en stor indsats på også at udvikle forskningsinfrastruktur på området, som efter flere års arbejde nu er på plads i Aarhus.
Denne udvikling glæder professor Poul Nissen: ”Med denne forskningsinfrastruktur og en stærkere og stærkere ekspertise inden for cryo-elektronmikroskopi er vi med helt fremme i verdenskonkurrencen om ny viden og muligheder på at forstå cellebiologi, lægemidler og bioteknologi på molekylært niveau. Det gør os i stand til at fastholde og udvikle Danmarks stærke position inden for strukturbiologi. Vi udvider snart mikroskopifaciliteterne med støtte fra infrastrukturprogrammet fra Ministeriet for Forskning og Innovation og kan dermed åbne for adgang fra forskningsgrupper på alle danske universiteter, industrien og start-up virksomheder. Det er af afgørende betydning for vores evne til at opdage nye mekanismer i samspillet af fysik, kemi, biologi og medicin og udvikle nye og innovative teknologier.”
Forskningen har været støttet af Forskningsrådet, Grundforskningsfonden og Ministeriet for Forskning og Innovation (nu Uddannelses- og Forskningsministeriet) samt Carlsbergfondet, Lundbeckfonden og Novo Nordisk Fonden. Milena har fået sine studier finansieret af et europæisk ph.d.-program fra Boehringer-Ingelheims Fond.
Link til den videnskabelige artikel i Nature: Structure and autoregulation of a P4-ATPase lipid flippase: Milena Timcenko1,5, Joseph A. Lyons1,5, Dovile Januliene2,5, Jakob J. Ulstrup1, Thibaud Dieudonne3, Cedric Montigny3, Miriam-Rose Ash1, Jesper Lykkegaard Karlsen1, Thomas Boesen1,4, Werner Kühlbrandt2, Guillaume Lenoir3, Arne Möller2 & Poul Nissen1
1DANDRITE, Nordic EMBL Partnership for Molecular Medicine, Department of Molecular Biology and Genetics, Aarhus University, Aarhus, Denmark. 2Max Planck Institute for Biophysics, Frankfurt, Germany. 3Institute for Integrative Biology of the Cell (I2BC), CEA, CNRS, Université Paris-Sud, Université Paris-Saclay, Gif-sur-Yvette, France. 4Interdisciplinary Nanoscience Center (iNANO), Aarhus University, Aarhus, Denmark. 5These authors contributed equally: Milena Timcenko, Joseph A. Lyons, Dovile Januliene.
Mere information
Professor Poul Nissen
Institut for Molekylærbiologi og Genetik/DANDRITE
Aarhus Universitet
pn@mbg.au.dk – 2899 2295
Ph.d.-studerende Milena Timcenko
milena@mbg.au.dk - 3013 4619
