Ny teknologi kaster lys over dynamiske RNA-protein-interaktioner
Mere end 80% af det menneskelige genom bliver transskriberet til RNA, der under transkribtionen bliver pakket med RNA-bindende proteiner (RBP'er). Dette enorme genomiske output giver en enorm udfordring - hvor og hvornår binder RBP'er sig til nylavet RNA for at dirigere dette til dets relevante eksport- eller nedbrydnings-veje? Dette spørgsmål addresseres nu af forskere fra Danmark og Skotland i et nyt Nature Communications studie.
Et samarbejde mellem professor Torben Heick Jensens og dr. Sander Grannemans laboratorier ved henholdsvis Aarhus Universitet og Edinburgh Universitet giver nu ny indsigt i den spatiotemporale dynamik af co-transskriptionel RNA-protein interaktion.
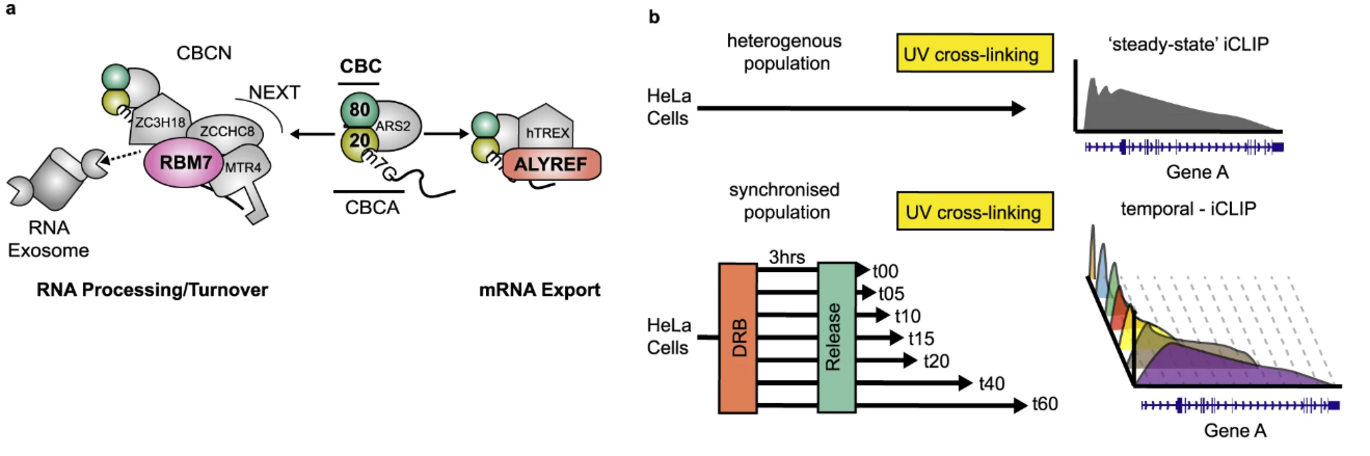
Den mest nøjagtige måde at studere sådanne interaktioner in vivo har været at inducere krydsbinding af RNA og protein i celler og herefter oprense RNA-protein komplekser og sekventere det krydsbundne RNA (såkaldt iCLIP teknologi). Dette involverer i det væsentlige bestråling af dyrkede celler med UV-lys for permanent at forbinde proteiner til de RNA'er, de interagerer med. Imidlertid kommer denne teknik til kort, fordi den kun fanger den gennemsnitlige protein-RNA-bindingsprofil for de celler, der dyrkes, men ignorerer dynamikken i protein-RNA-bindingerne.
Udvikling af en ny teknik – tiCLIP
For at imødekomme denne udfordring udviklede Cordiner et al. en teknik kaldet temporal iCLIP (tiCLIP), som måler protein-RNA-interaktioner i et snævert tidsopløst vindue efter genstart af transskription, efter dens forudgående nedlukning. Til analysen anvendte forskerne RNA-eksportfaktoren ALYREF, RNA-nedbrydningsfaktoren RBM7 og de cap-bindende komplekskomponenter CBP20 og CBP80 (se figur).
tiCLIP teknikken viser, at uanset hvilken rolle de pågældende proteiner spiller i RNA metabolismen, så interagerer de promiskøst med det nydannede RNA, så snart det forlader transskriptionsmaskineriet. Disse tidlige interaktioner udgør imidlertid en indledende fase af forbigående RBP-RNA interaktioner, der senere erstattes af stærkere affinitetsforankringssteder.
Mere detaljeret klargør resultaterne, at mRNA-splejsningsprocessen midlertidigt adskiller binding af RNA-eksport- og -nedbrydnings-faktorer, der binder til forskellige splejsningsmellemprodukter.
Slutteligt afslørede den høje spatiotemporale opløsning opnået ved tiCLIP teknologien, hidtil ukendte intermediater af små nukleolære RNA (snoRNA'er) bundet til RNA-henfaldsfaktoren, RBM7, som nu kan bruges til at kaste lys over den mekanisme, som færdigudvikler snoRNA molekyler.
Forskerne arbejder nu på en videre optimering af tiCLIP teknologien og at applicere den på flere RBP’er. Et vigtigt mål er også, at opnå en opløsning, hvor CLIP kan bruges til at måle RBP-RNA binding direkte i vævsprøver.
SUPPLERENDE OPLYSNINGER, HERUNDER KONTAKTOPLYSNINGER
Vi bestræber os på, at alle vores artikler lever op til Danske Universiteters principper for god forskningskommunikation. På den baggrund er artiklen suppleret med følgende oplysninger:
PUNKTER | INDHOLD OG FORMÅL |
Studietype | Eksperiment |
Ekstern finansiering | Dette arbejde er blevet støttet af the Lundbeck and Novo Nordisk foundations (NNF, ExoAdapt Grant 311, an EMBO long-term fellowship (ALTF 1070-2017) and a Marie Curie Individual Fellowship (797358), Medical Research Council Non-Clinical Senior Research Fellowship (MR/R008205/1). |
Interessekonflikt | Ingen |
Link til den videnskabelige artikel | Ross A. Cordiner, Yuhui Dou, Rune Thomsen, Andrii Bugai, Sander Granneman & Torben Heick Jensen Temporal-iCLIP captures co-transcriptional RNA-protein interactions Nature Communications https://doi.org/10.1038/s41467-023-36345-y
|
Kontakt- information | Professor Torben Heick-Jensen |
