Et nyudviklet RNA-molekyle binder til SARS-CoV-2 virus og blokerer dets optag i celler
Et forskerhold fra Institut for Molekylærbiologi og Genetik og iNANO på Aarhus Universitet har brugt en in vitro selektionsteknik til at udvikle et RNA-molekyle, der genkender og binder Spike-proteinet fra coronaviruset SARS-CoV-2 med meget høj specificitet og styrke. Håbet er, at dette nye molekyle, også kaldet en aptamer, kan bidrage til bedre diagnostik og behandling af COVID-19 på længere sigt.
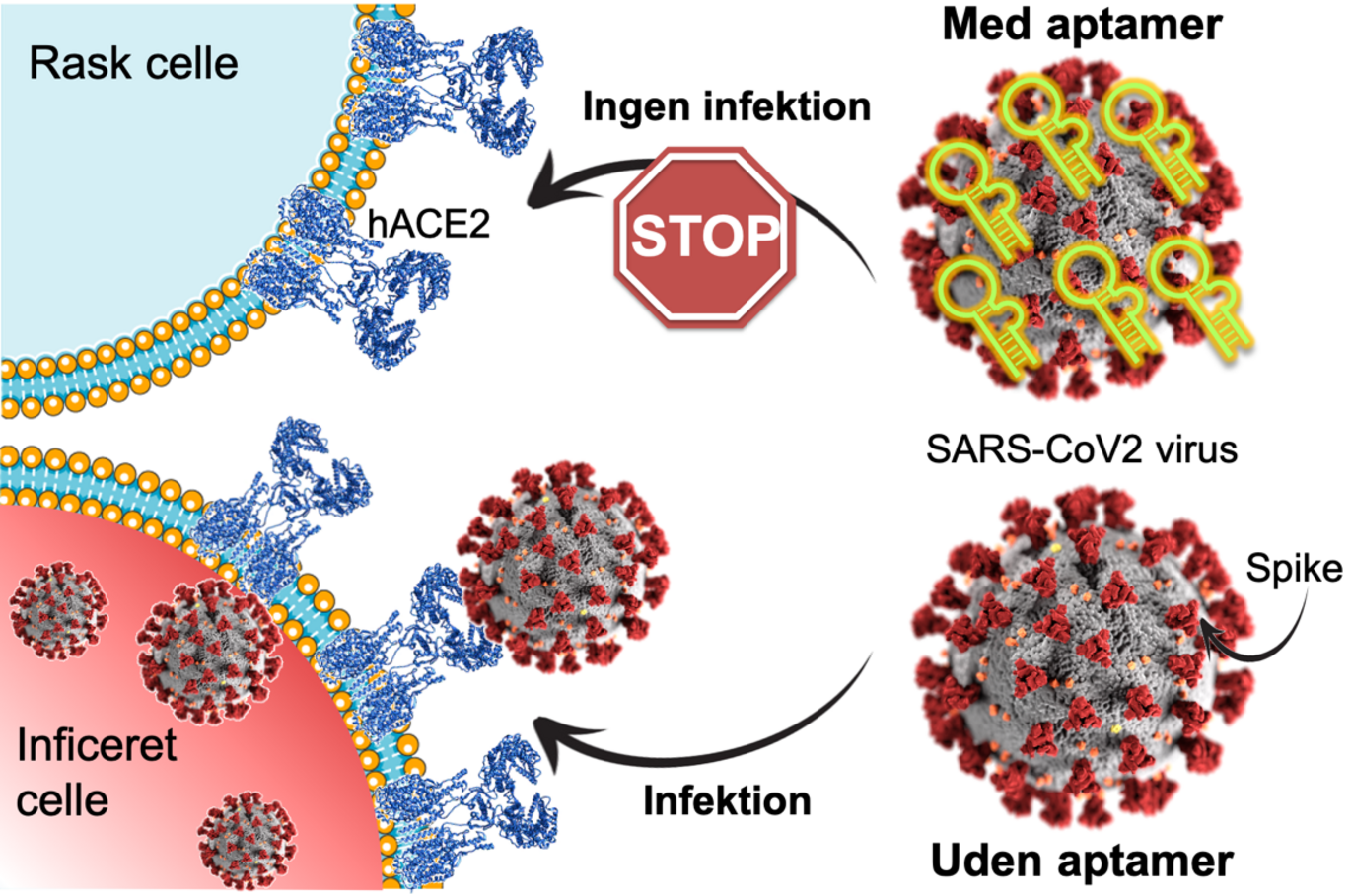
Det seneste halvandet års COVID-19-pandemi har understreget vigtigheden af at udvikle nye molekyler til at diagnosticere og bekæmpe virussygdomme. En ny forskningsartikel i PNAS fra Julián Valero, Laia Civit, Jørgen Kjems samt kollegaer i Aarhus og Regensburg i Tyskland beskriver udviklingen af en RNA-aptamer med meget høj bindingsaffinitet for Spike-proteinet lokaliseret på overfladen af SARS-CoV-2 viruspartikler. Ved at klistre sig fast på virusoverfladen kan aptameren forhindre, at Spike-proteinet bruges som en nøgle, der gør det muligt for virus at trænge ind i cellerne. RNA-aptameren folder sig i en 3D-struktur, som gør det muligt at genkende et specifikt molekyle. På den måde kan aptamerer sammenlignes med traditionelle antistoffer, men de er langt nemmere og billigere at fremstille.
Det nye studie viser, at aptameren effektivt blokerer bindingen af Spike-proteinet til receptoren ACE2 på menneskeceller og derved effektivt neutraliserer infektion af humane celler i cellekultur. Desuden viser studiet, at RNA-aptameren virker mod de førstbeskrevne varianter af coronavirus med samme effektivitet. “Siden har vi arbejdet videre og vores undersøgelser viser, at aptameren også genkender delta-varianten. Nu venter vi på eksemplarer af den nyfundne variant, omikron, så vi kan teste aptameren også kan genkende den,” siger Jørgen Kjems.
De lovende resultater peger på en mulig fremtidig behandling af COVID-19 samt udviklingen af billigere og mere sensitive diagnostiske hurtigtest. Julian Valero, som er førsteforfatter på artiklen, understreger at teknikkens brede potentiale inden for virusdetektion: 'Vi kan bruge teknologien til at udvikle nye RNA aptamerer, der kan rettes mod fremtidige varianter af SARS-CoV-2 og mod andre sygdomsfremkaldende virus.'
Forskningsprojektet er støttet af Carlsbergfondet og af EU FET-open-projektet VIROFIGHT. Jørgen Kjems og Julián Valero er derudover begge medlemmer af CellPAT, et Center of Excellence støttet af Danmarks Grundforskningsfond.
Kontaktdata
Professor Jørgen Kjems, Institut for Molekylærbiologi og Genetik samt Interdisciplinary Nanoscience Center (iNANO), Aarhus Universitet, jk@mbg.au.dk, 2899 2086
Adjunkt Julián Valero, Interdisciplinary Nanoscience Center (iNANO), Aarhus Universitet, jvalero@inano.au.dk
Ekstern finansiering: Carlsbergfondet bevillingsnr. CF20-0045, Danmarks Grundforskningsfond bevillingsnr. DNRF135, EU Horizon2020 bevillingsnr. 899619
Interessekonflikt: Forskerne har ingen interessekonflikt
Andet: Aarhus Universitet har indsendt en patentansøgning for den udviklede RNA-aptamer
Valero J*, Civit L,* Dupont DM, Selnihhin D, Reinert LS, Idorn M, Israels BA, Bednarz AM, Bus C, Asbach B, Peterhoff D, Pedersen FS, Birkedal V, Wagner R, Paludan SR, Kjems J: A serum-stable RNA aptamer specific for SARS-CoV-2 neutralizes viral entry. PNAS December 14, 2021 118 (50) e2112942118; doi.org/10.1073/pnas.2112942118.
