Ny sekventeringsmetode afslører kompleksiteten af cirkulære RNA-molekyler i hjernen
Jørgen Kjems' forskningsgruppe beskriver i en artikel i <em>Nature Communications</em>, hvordan de har udviklet en ny metode til at bestemme sekvensen af en klasse af RNA-molekyler kaldet cirkulært RNA (circRNA). Undersøgelser i hjerneprøver fra mennesker og mus viser, at circRNAer findes i mange forskellige versioner, og at en særlig klasse af genetiske elementer med en mulig rolle i autisme oftere findes i circRNA end i de mere hyppige lineære mRNAer.
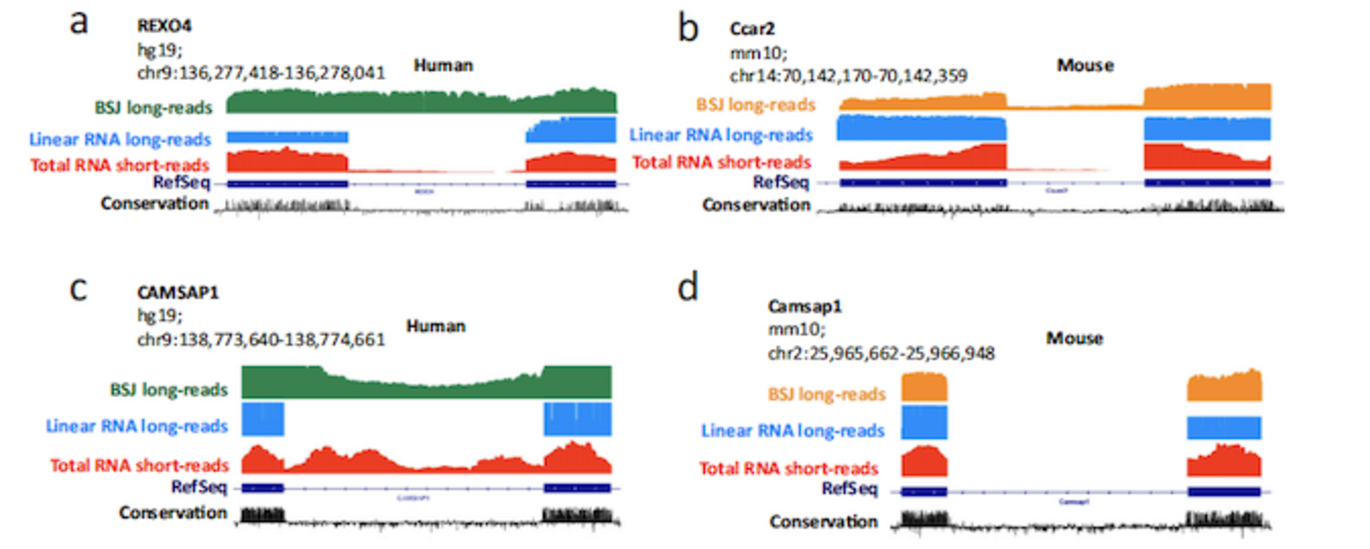


Når celler læser den information, der ligger i vores gener, laver de først en RNA-kopi af det enkelte gen. RNA-molekyler forarbejdes på et utal af måder, hvilket skaber den kompleksitet i gen-udtrykket man ser i højere organismer. Det gennemsnitlige gen producerer syv forskellige RNA isoformer via alternativ splejsning, og denne proces er særlig aktiv i neuroner. Cirkulære RNAer (circRNAer) er en gruppe af meget stabile RNA-molekyler, der er hyppige i neuroner, men deres interne sammensætning er relativt uudforsket, da de overlapper med det mere velkendte lineære mRNA.
En af udfordringerne for at forstå cirkRNAernes sammensætning er, at de hidtil kun er blevet beskrevet ved hjælp af sekventeringsteknikker, der læser korte strækninger af RNA. Jørgen Kjems' gruppe har løst dette problem ved at udvikle protokoller, hvor cirkRNAer først isoleres og derefter bliver analyseret ved hjælp af Nanopore sekventeringsteknologi, hvor mange lange stykker RNA kan læses på en gang.
Ud over et sæt ny laboratorieprotokoller indeholder studiet også en ny bioinformatisk platform for dataanalyse, som er udviklet af tidligere MBG/INANO-postdoc Morten Venø, nu administrerende direktør for AU spin-out-selskabet Omiics. Samlet set har den nye metode ført til den overraskende opdagelse, at circRNAer udviser mere forskelligartede splejsning end lineært mRNA.
Forfatterne opdagede desuden, at en særlig gruppe af meget korte exons, de såkaldte mikroexons, oftere findes i circRNAer end i lineære RNAer. Flere af disse mikroexons er tidligere blevet forbundet med autisme, hvilket tyder på, at den unikke sammensætning af circRNAer i hjernen kan spille en rolle i hjernesygdomme.
Artiklen i Nature Communications:
"Nanopore sequencing of brain-derived full-length circRNAs reveals circRNA-specific exon usage, intron retention and microexons"
Karim Rahimi, Morten T. Venø, Daniel M. Dupont, Jørgen Kjems
DOI: 10.1038/s41467-021-24975-z
Mere information
Jørgen Kjems
iNANO/Institut for Molekylærbiologi og Genetik, Aarhus Universitet
jk@mbg.au.dk – 2899 2086
Karim Rahimi
iNANO/Institut for Molekylærbiologi og Genetik, Aarhus Universitet
karim@mbg.au.dk
Morten Venø
Omiics
morten.venoe@omiics.com
