Torben Heick Jensens gruppe ved Institut for Molekylærbiologi og Genetik, Aarhus Universitet, har opdaget en ny mekanisme, der sikrer, at ikke-produktivt RNA nedbrydes effektivt i stress-påvirkede celler. Ved at sætte kæder af nukleotiderne adenin og uridin på RNA’ets 3’-ende kan cellen sikre, at ikke-produktivt RNA bliver nedbrudt, hvis det kommer uden for cellekernen.

Forskere fra Institut for Molekylærbiologi og Genetik har i samarbejde med andre forskere opdaget en overraskende mekanisme for, hvordan RNA Polymerase II (RNAPII) afslutter transskription i eukaryote celler (med cellekerne). Det viser sig at kunne ske via simple DNA-sekvenser; kaldet T-tracts. Denne mekanisme minder til forveksling om den, der anvendes af RNA Polymerase III (RNAPIII) og af RNA polymerasen i prokaryote celler (uden cellekerne). Opdagelsen peger således på en bredere og evolutionært bevaret rolle for T-tracts i den afsluttende transskriptions proces.

Forskning fra Torben Heick Jensens gruppe ved Institut for Molekylærbiologi og Genetik tilføjer et nyt lag af kompleksitet til selektiv RNA-nedbrydning. Forskerne har opdaget et adaptor-kompleks, vNEXT, som genkender RNA-molekyler baseret på deres foldning frem for deres sekvens.

Når vores gener transkriberes til RNA, sker der ofte fejl tidligt i processen. I en særudgave af tidsskriftet Molecular Cell med fokus på RNA-biologi diskuterer William Garland og Torben Heick Jensen fra MBG de tidlige mekanismer for kvalitetskontrol, som sikrer den rette sammensætning af RNA-molekyler i vores celler.
Transskription er den proces, hvorigennem DNA kopieres til RNA. Overraskende nok når kun et fåtal af transskriptionsforløbene at blive tilendebragt, sandsynligvis på grund af effektiv kvalitetskontrol af transskriptionsreaktionen. Nye resultater viser nu en ny måde, hvorpå dette kan ske.
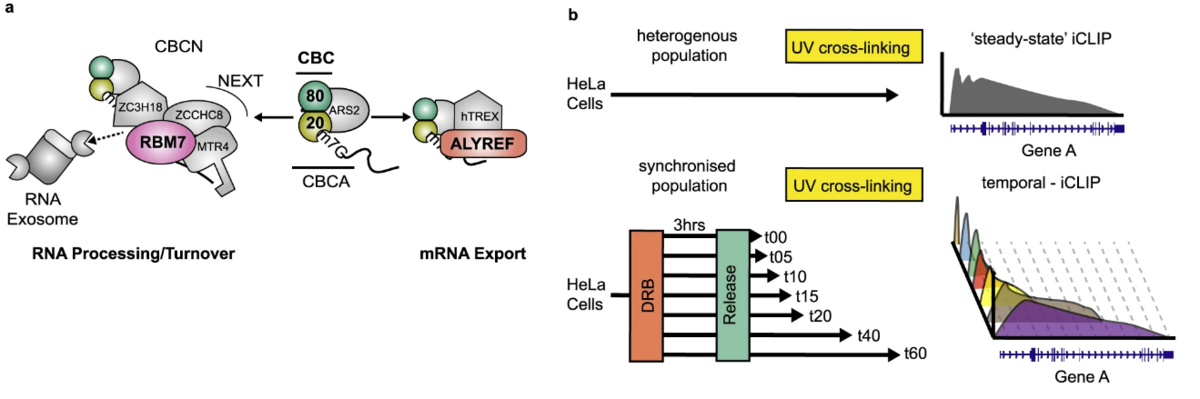
Mere end 80% af det menneskelige genom bliver transskriberet til RNA, der under transkribtionen bliver pakket med RNA-bindende proteiner (RBP'er). Dette enorme genomiske output giver en enorm udfordring - hvor og hvornår binder RBP'er sig til nylavet RNA for at dirigere dette til dets relevante eksport- eller nedbrydnings-veje? Dette spørgsmål addresseres nu af forskere fra Danmark og Skotland i et nyt Nature Communications studie.
Forskere fra Aarhus, Cambridge og Warszawa har i et fælles forskningsprojekt karakteriseret, hvordan de vigtige mRNA poly(A)-haler syntetiseres i bagegær. Undersøgelsen afslører overlappende mekanismer, der styrer poly(A)-halens længde og hermed sikrer stabiliteten af cellens genekspression.
Produktion af RNA i cell-kernerne i eukaryote organismer er under kontrol af det nukleære RNA-exosom hjulpet af adskillige co-faktorer. Forskere ved Aarhus Universitet og Københavns Universitet viser nu, hvordan to vigtige co-faktorer af det nukleære exosom genkender deres RNA-targets for at bevare en korrekt RNA-balance i cellekernen. Dette er vigtigt for cellernes – og dermed menneskers – overlevelse.
Forskere fra Aarhus Universitet og Københavns Universitet viser i et nyt studie, at for meget RNA i cellekernen kan have negative konsekvenser for afgørende regulator af stamcelle-differentiering.
Et centralt spørgsmål i biologien er, hvordan celler oversætter deres genetiske information til funktionelt RNA og protein. Forskere fra Institut for Molekylærbiologi og Genetik på Aarhus Universitet har udviklet en ny teknologi til at studere reguleringsprincipper for genekspression.
Ved at anvende denne metode på bagegær fandt de, at RNA'er, der ikke eksporteres fra deres syntesested i cellekernen, hurtigt nedbrydes. Dette giver et nyt reguleringsprincip i den komplekse proces af genekspression.
Forskere ved Aarhus Universitet har karakteriseret den humane RNA nedbrydningsfaktor kaldet ZC3H18 og opdaget en uventet rolle i produktionen af proteinkodende RNA. De nye resultater, der blev offentliggjort i denne uge i Cell Reports, afslører således en tvedelt aktivitet af ZC3H18 i beslutninger om nukleær RNA-skæbne.
Vores genomer transskriberes promiskuøst til RNA. Hvordan celler formår at sortere dette massive genomiske output i funktionelt og ikke-funktionelt materiale har været en gåde. Ny forskning beskriver nu protein-interaktioner, der er med til at bestemme RNAs skæbne.
Genomer transskriberes til et hav af forskelligt RNA. Dette materiale er ikke alt sammen anvendeligt, hvorfor målrettet nedbrydning er essentielt for at opretholde cellulært liv. En nyligt opdaget RNA nedbrydningsvej i menneskets cellekerner er med til netop det.
Intense studier de seneste 10-15 år har vist, at det humane genom transskriberes på en måde, der er langt mere kompliceret, end man tidligere troede. Et samarbejde mellem forskere fra Aarhus og København afslører nu nogle underliggende principper, der fører til sådan varieret genomaktivitet.
Nye forskningsresultater kortlægger funktionen af et protein, der spiller en vigtig rolle i nerveceller. Yderligere undersøgelser af mekanismen kan føre til en forståelse af, hvordan skader på nerveceller opstår, når proteinet er muteret.
I samarbejde med to andre europæiske grupper har forskere fra Aarhus Universitet afdækket de molekylære detaljer, der fører til målrettet rekruttering af det vigtige nedbrydningsmaskineri, RNA exosomet, til dets nukleare RNA substrater. Fremtidige undersøgelser kan nu afdække, om denne RNA nedbrydningsvej er kritisk under embryonal udvikling, celledifferentiering og forskellige former for stress.
Professor Torben Heick Jensen fra Institut for Molekylærbiologi og Genetik, Aarhus Universitet, har modtaget en Semper Ardens Advance-bevilling på 25 millioner kr fra Carlsbergfondet til at undersøge de molekylære processer, der nedbryder ikke-funktionelle og potentielt skadelige RNA-molekyler i cellekernen.
Professor Torben Heick Jensen, Institut for Molekylærbiologi og Genetik, Aarhus Universitet, modtager 60 mio. kr. fra Novo Nordisk Fondens Challenge Program til at etablere forskningscenteret ’Exo-Adapt’, der skal beskrive, hvorledes vore celler sorterer genetisk information.

På medlemsmødet den 31. marts 2016 i Det Kongelige Danske Videnskabernes Selskab blev Professor Torben Heick Jensen indvalgt som medlem til den Naturvidenskabelige Klasse.
Aktiviteten af det menneskelige genom er enorm. Således bliver 70-80% af dets DNA transskriberet til RNA, og forskere verden over har gennem mange år forsøgt at finde ud af, hvor meget af dette materiale, der er funktionelt. Torben Heick Jensen har nu fået en prestigefyldt bevilling fra Lundbeckfonden til at undersøge.
eLife er et peer-reviewed videnskabeligt tidsskrift for biomedicin og biovidenskab med open access. Det blev sponsoreret af Howard Hughes Medical Institute, Max Planck Society og Wellcome Trust efter en workshop afholdt i 2010 på Janelia Farm Research Campus, og i slutningen af 2012 blev det officielt etableret.
Professor Torben Heick Jensens forskning i genomekspression, stabilitet og teknologi bliver nu støttet af en Advanced Grant fra Det Europæiske Forskningsråd.
Torben Heick Jensen – professor ved Institut for Molekylær Biologi og Genetik og leder af Center for mRNP Biogenese og Metabolisme finansieret af Dansk Grundforskningsfond – er inviteret til at holde et foredrag i den prestigefyldte ‘Mendel Lecture series’. Arrangementet finder sted den 18. april 2013 i Brno, Tjekkiet.
Professor Torben Heick Jensen fra Institut for Molekylærbiologi og Genetik ved Aarhus Universitet har opnået stor international anerkendelse ved valget som medlem af den europæiske molekylærbiologiske organisation EMBO (the European Molecular Biology Organization). Valget til EMBO er begrundet i Torben Heick Jensens fremragende forskning inden for genekspression.
The Centre for mRNP Biogenesis and Metabolism studies the crosstalk between transcriptional and post-transcriptional processes, which establishes a network of interdependencies that ultimately regulate gene expression. Our focus is on structure/function relationships of mRNP formation and its quality control as well as the occurrence, and putative function, of non-coding RNA transcription.